Відкритий лист: щодо плану магістерської програми з біоінформатики в ПНУ ім. Василя Стефаника
Ви могли побачити новини, що Володимир Швадчак з колегами запускають нову магістерську програму з біоінформатики в ПНУ ім. Василя Стефаника. Персонально, мені імпонує цей заклад і особливо кафедра біохімії, команда якої гарно публікує результати своїх досліджень. Щобільше, вони зробили те, що, на мою думку, повинен робити будь-який заклад освіти: винесли на громадське обговорення проєкт магістратури і активно залучають до цього фахівців, колег та майбутніх студентів. Як фахівець з біоінформатики та обчислювальної біології, я радий взяти в цьому участь і запрошую вас. Нижче я залишаю свій текст як відкритий лист, щоб, по-перше, потім передивитися як розвиватиметься програма, а по-друге, якщо це допоможе іншим колегам у майбутньому під час розробки їх програм. А саме, два пункти позитивної критики для того, щоб, на мою думку, ця програма стала кращою.
1. Кадри
На мою думку, інші освітні програми з біоінформатики в Україні не є повноцінними та якісними через те, що в їх викладацькому складі зазвичай багато профнепридатних людей. Якщо фахівець не публікується раз на декілька років хоча б в журналі рівня BMC Bioinformatics, або не має регулярного комерційного/core facility/bioinformatics core досвіду як біоінформатик або консультант (індустрія/стартап) - така людина не може викладати на курсі з біоінформатики, її місце на касі в МакДональдз. На жаль, якщо хоча б одна така людина потрапить до викладацького складу магістерської програми (не зважаючі на публікації в мурзилках з переліку МОН, вельмишановність або титулованість) - цю програму можна вважати невдалою.
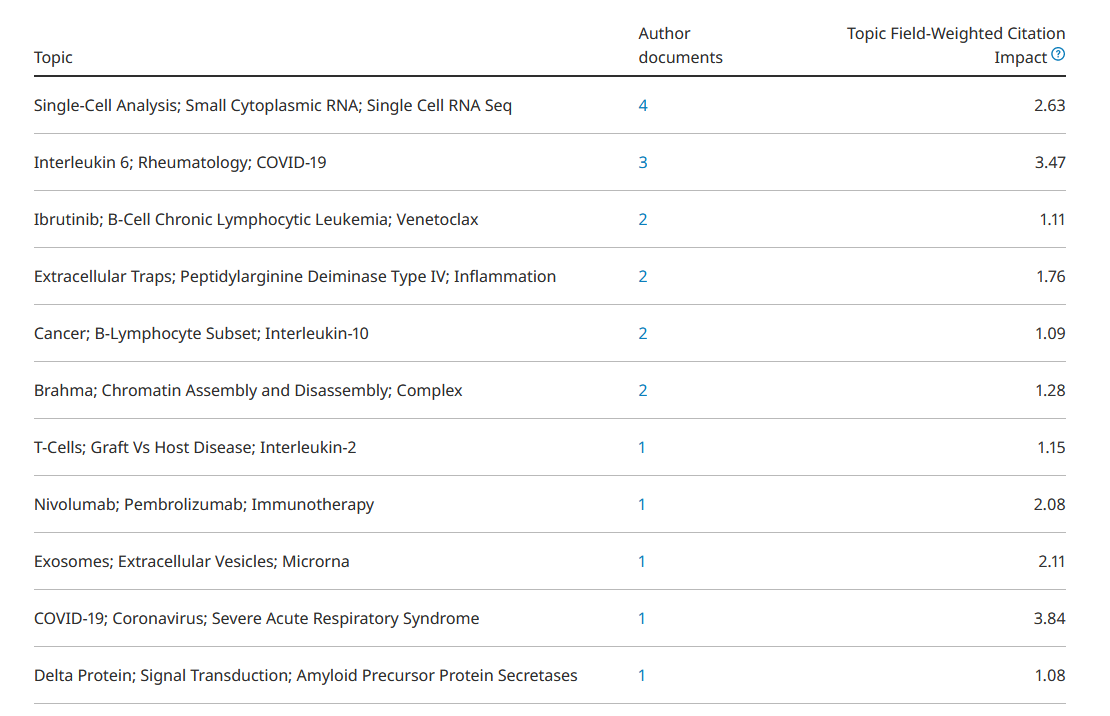 Приклад: профіль біоінформатика з досвідом, потрібним для викладання на магістерській програмі. Джерело: Scopus, де topic field-weighted citation impact > 1 показує, що автора цитують більше, ніж в середньому у сфері .
Приклад: профіль біоінформатика з досвідом, потрібним для викладання на магістерській програмі. Джерело: Scopus, де topic field-weighted citation impact > 1 показує, що автора цитують більше, ніж в середньому у сфері .
Я подивився наукові профілі робочої групи, яка працювала над створенням проєкту. Не зважаючи на те, що наукові здобуття деяких її представників солідні не лише за українськими, а і за світовими мірками, я не побачив там фахівців з біоінформатики. Чи готова ця робоча група зайнятися рекрутингом, а потім відійти від власної програми, або вона, скоріше, бачить це як “learn by doing” зі суміжних спеціальностей стосовно себе? Я не впевнений, що останній підхід є життєздатним на тому рівні, який очікується від магістерських програм.
З гарних новин: ця проблема не є унікальною для України, і у світі майже нема країн, які завдяки “внутрішнім” кадрам можуть закрити усі необхідні faculty-level позиції. Для цього існує а) міжнародний рекрутинг та б) так звані “adjunct”-позиції.
а) Складно зрозуміти, скільки людей з релевантним досвідом захочуть зараз релокуватися до України. До того ж мені невідомо, чи здатний Прикарпатський університет конкурувати за компенсацією зі, скажімо, аналогічними позиціями в інститутах Чехії або Польщі (а якщо ні - то чи може він дозволити собі нову магістерську програму, де викладати повинні фахівці, кваліфікація яких апріорі коштує дорого?). Проте, є гіпотетична ймовірність залучити українців на початковому рівні кар’єри (постдоки+), які хотіли б продовжити працювати вдома.
б) Щодо adjunct-позицій, це простіше: на таку позицію запрошується фахівець, який продовжуватиме працювати за основним місцем роботи, проте паралельно викладає або проводить дослідження на вашій програмі - чи то в дистанційному форматі, чи приїжджаючи на конкретні курси. Скоріше за все, на таку позицію можливо залучити навіть враховуючи звичайну компенсацію у Прикарпатському університеті, тобто фактично pro bono, через те, що як фахівці-українці, так і іноземні колеги, що співчувають нам, були б не проти допомогти.
2. Програма.
Я з великим задоволенням перечитав пункти, які запропонував у своєму дописі пан Володимир. Це - дуже потужний і близький погляд біолога на те, як може виглядати освітня програма з біоінформатики. Проте, якщо ультимативна мета - щоб випусники отримували кар’єрні можливости десь поза закладами НАНУ, я думаю, що її треба модифікувати.
Перед тим, як перейти до деталей, я пропоную читачу зробити просту вправу. Відкрийте LinkedIn і введіть там bioinfmatician в пошуку - окремо для Європи і окремо для Північної Америки. Перше, що ви побачите - що більшість вакансій вимагає MSc + досвід АБО PhD. Тобто, очікується, що ваша програма готуватиме фахівців, які можуть конкурувати на ринку з випускниками ETH Zurich, UCL та MIT - а їм доведеться, оскільки на одні й ті самі вакансії подаються десятки і сотні людей з усього світу. Друге, що ви побачите - вимоги і необхідні компетенції. І я пропоную розглядати програму саме через призму них.
Наприклад, пан Володимир приводить, що випускники працюватимуть в лабораторіях, які займаються аналізом геному та динаміки поширення вірусів. На жаль, це дуже нішеві напрями, які обмежують можливості для роботи. Повертаючись до LinkedIn, ви побачите, що, умовно, усі вакансії для біоінформатиків можна розділити на структурні/CADD (мало і, можливо, не ваш профіль?), software-орієнтовані (мало і не ваш профіль) та оміксні (більшість). В останніх ви побачите найбільший запит на таких, які займаються речами на кшталт мультиомік, CRISPR/drug screens, imaging analysis, spatial biology.
 Приклад: перші результати пошуку за запитами Bioinfomatician / European Union”.
Приклад: перші результати пошуку за запитами Bioinfomatician / European Union”.
Стратегічна проблема в тому, що, якщо фахівець у 2024 році вміє лише працювати з геномними та протеомними даними, як зазначено у дописі Володимира (дивний фокус/комбінація, відверто), то він/вона фактично не здатні конкурувати на ринку. Бо що геномні дані, що протеомні сьогодні аналізують за допомогою pipelines; щобільше, якщо лабораторія сфокусована на геномних даних, то, скоріше за все, в них є доступ до комерційних систем від виробників обладнання (див. Illumina BaseSpace, Illumina DRAGEN), які роблять більшість рутиного аналізу таким, з яким здатні працювати technical assistant (TA) з бакалавратом. Те ж саме стосується і протеоміки: на мою персональну думку, з усієї мультиоміки, саме спектральні дані (LC/MS proteomics, metabolomics) є найменш складною проблемою для аналізу. Щобільше, зазвичай в core facilities первинну обробку даних та навіть певний downstream здатні зробити ті ж самі TA, бо для цього використовують готові рішення на кшталт ThermoFisher Protome Discoverer; якщо ж в людей є бажання працювати самостійно з матрицями спектральних прочитань і робити власний аналіз, як раджу робити студентам я, то для цього не треба півтора року навчатися на магістратурі - це здатен зробити й бакалавр з computer science. До того ж я не зустрів у проєкту програмі жодної згадки того, що вже дуже багато років є “королем” омік - bulk RNA-seq і більш сучасних scRNA-seq/snRNA-seq у різних модифікаціях. На відміну від протеоміки, про це як раз можна розмовляти півтора року (а краще разом з протеомікою в контексті інтеграції), бо це, по-перше, є найбільш затребуваним в академії, по-друге, відкриває шлях до spatial biology, яка як раз буде на пику коли ваші перші випускники отримують ступені, по-третє, використовується майже в кожній біофарм-компанії.
У програмі, яку підсумував пан Володимир, відведено 30% на програмування/інформатику, 30% на загальні навички, і аж 40% на біологію. Тут є декілька проблем. По-перше, я не впевнений, чи ви хочете робити біоінформатиків з тих біологів, які не мають жодної підготовки з computer science, хоча б в межах неформальної освіти. Навчати людей як працюють функції в Python (чомусь пріоритет відданий йому) - це не завдання магістерських програм. І навпаки, вашими слухачами можуть бути люди з підготовкою у computer science. Відповідно, має сенс винести усі зайняття з Unix/Bash/SQL/Python/R/Julia/Rust/інше у факультатив, й зробити цей факультатив інтенсивним курсом прямо на старті програми. А ось що треба додати обов’язково - алгоритми (будь ласка, не моделювання, як написано в програмі - це дуже сильно різні речі). До того ж у вашій програмі зовсім нема нічого з networks, causal inference, machine learning, manifolds, dimensionality reduction, experimental design - будь ласка, зробіть ревізію цієї частини, без розуміння цього (і без вказання у CV) вашим випускникам буде дуже важко знайти роботу.
По-друге, на біологію відведено аж 40% часу, з 3 кредитами та екзаменом (!) з ПЛР та секвенування, куди також чомусь віднесена підготовка бібліотек. Якщо вам хочеться розповісти біоінформатикам про ПЛР та підготовку бібліотек - вставте про це один слайд на вступній лекції програми. Це - те, чим НЕ займаються біоінформатики, і окрім як “добре знати” не є релевантним. Випускник цієї спеціальності або це вже знає, або ніколи не буде працювати в wet lab умовах. Також, рекордні 6 кредитів відведені на філогенетику і молекулярну еволюцію - див. пораду з LinkedIn, це не є вирішальною навичкою, яка допоможе у працевлаштуванні вашим студентам.
Щодо soft skills/загальних навичок - на мою думку, це дуже гарний напрям і те, чого зазвичай нема в українських університетах. Я сподіваюся, що основи підприємництва читатимуть лектори з Enamine/Receptor.AI/Explogen, а не представники стагнуючих стартапів та компаній.
Резюме
Програма, про яку йдеться в обговорені, потрібна для українського ринку освіти. Проте, у наявній формі, без залучення фахових біоінформатиків, є високий ризик, що вона опиниться на цвинтарі освітніх програм - там же, де вже знаходиться решта українських програм з біоінформатики. Програмі потрібна масштабна ревізія щодо освітньої програми і введення “важких” об’єктивних фільтрів стосовно того, хто може на ній викладати, а хто - ні. Враховуючи реалії українського освітнього ринку, чи не єдиною запорукою успіху є колаборації з тими закладами та людьми, в яких є необхідний вам досвід. Наприклад:
1) Партнерство з УКУ Data Science програмою для ревізії вашої computer science частини та залучення їх фахівців до викладання.
2) Партнерство з КАУ щодо успішного досвіду “практичних” магістратур, які найкраще готують студентів до ринку праці (приєднання до їх програм або залучення до програми).
3) Партнерство з вченими, в тому числі українського походження, які мають підтверджений великий досвід в біоінформатиці й могли б викладати на вашому курсі як adjunct-faculty. Наприклад, Аліна Фролова, Тарас Олексик, Сергій Науменко, Serghei Mangul, Олексій Рухленко, Ростислав Кузяків. Врешті-решт, з нами як найбільшою організацією з біоінформатики та омік в Україні (ГО “Геноміка ЮА”).
До того ж ваші студенти можуть виграти від повного переведення програми на англомовну з залученням міжнародних учасників faculty. Бажаю вам успіхів і буду радий, якщо мій погляд допоможе вам удосконалити програму.



